1. CRISPRiによりバクテリアにおけるMGE調節
[出典] "CRISPR-interference based modulation of mobile genetic elements in bacteria" Nyerges A, Balint B, Cseklye J, Nagy I, Pal C, Feher T. bioRxiv. 2018-09-27.
- 合成生物学においてバクテリアに導入する合成遺伝子は、可動遺伝子因子群 (MGEs)が誘導する突然変異によって、設計した機能を短時間で失う。この問題の解決にこれまでは、バクテリア染色体の編集と全ての挿入配列 (ISes)を削除するという極めて長時間を要する操作を要していた。
- ハンガリーの Institute of Biochemistry, Biological Research CentreとSeqomics Biotechnology Ltd.の研究チームは今回新たな問題解決の手法として、CRISPRiによってバクテリアのISesの転移を阻害する単一プラスミド・システム (a single plasmid-based system, pCRIS)を構築した。
- pCRISは、 Escherichia coli in vivoで、38遺伝子座に存在するIS1, IS3, IS5およびIS150の同時サイレンシングを可能とする多重gRNAsを発現する。これによって、染色体とエピソームの双方において標的の4ISesの転移レベルが無視できる程度まで低減し、外来遺伝子からのタンパク質発現の半減期が伸びた。pCRISの効力は、ゲノム・スケールでの染色体工学による操作と同等であった。
- pCRISは多様なE. coli菌株において転移サイレンシングを実現した。
2. Lipofectamineトランスフェクション試薬を利用することで、非モデル線虫におけるRNAiとCRISPRを介した変異誘発を亢進する
[出典] "Liposome-based transfection enhances RNAi and CRISPR-mediated mutagenesis in non-model nematode systems" Adams S, Pathak P, Shao H, Lok J, da Silvabio AP. bioRxiv. 2018-09-27.
- モデル生物に有効な機能解析手法はしばしば非モデル生物には有効ではない。 U. WarwickとU. Pennsylvaniaの英米の研究チームは今回、2017年に記載されたAuanema属線虫 の耐久型幼虫にも遺伝性変異を導入可能な手法を開発し、Pristionchus pacificus とヒト寄生Strongyloides stercoralisへのRNAiにも有用なことも示した。
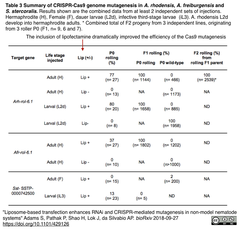
- 本手法では、Lipofectamineトランスフェクション試薬とdsRNAまたはCRISPR Cas9 RNP-ssDNAとの混合液をマイクロインジェクションする (lipofectamineの効果、原論文Table 3引用下図参照)。
3. FLASH: 薬剤耐性 (antimicrobial resistance, AMR)遺伝子検出を多重化する次世代CRISPR診断法
[出典] "FLASH: A next-generation CRISPR diagnostic for multiplexed detection of antimicrobial resistance sequences" Quan J, Langelier C, Kuchta A [..] Crawford ED. bioRxiv. 20198-09-27.
- 抗生物質に対する耐性を獲得した病原微生物はヒトの健康に対する脅威である。臨床分離培養株からのAMR遺伝子検出は容易であるが、臨床検体の高いバックグラウンドから低含量のAMR遺伝子を直接検出することは容易ではない。
- UCSF, Stanford U, Sanger Inst.ならびにNamibia Uの研究チームは今回、FLASH (Finding Low Abundance Sequences by Hybridization)法を開発し、臨床検体からkのAMR遺伝子の直接検出を実現した。
- FLASHでは、ゲノムDNAまたはcRNAを脱リン酸化により再結合をブロックし、Cas9とAMR遺伝子を標的とするgRNAsセットでイルミナ・シーケンシングに適したサイズにまで消化した上で、アダプター連結、増幅を経て、バックグラウンドに対してAMR遺伝子断片を濃縮し、NGSで解析する。
- CARDとResFinderデータベースからAMR関連遺伝子3,624遺伝子配列を抽出し、5,513 gRNAsを設計し、127 AMR遺伝子で、臨床分離株と患者4名からの気道液および乾燥血液スポット (Dried Blood Spot, DBS)で検証した。
- FLASH-NGSはNGSに対して濃縮度が平均5,000倍 (563~13,244倍)に達し、バックグラウンドを最小限にとどめ、濃度アトモル未満の遺伝子検出を実現した。また、gRNA設計用ソフトウエアツールFLASHitを公開した。
コメント