2016年05月10日
CRISPR関連文献メモ_2016/05/10(1件)
[crisp_bio 注] 本論文には出版直後から批判が続き、Nature Biotechnologyからリトラクトされるに至った:2017-08-04投稿「アルゴノート(NgAgo)ってどうよ?(6)終幕」を参照
CRISPR関連文献メモ_2016/05/10(1件)
[crisp_bio 注] 本論文には出版直後から批判が続き、Nature Biotechnologyからリトラクトされるに至った:2017-08-04投稿「アルゴノート(NgAgo)ってどうよ?(6)終幕」を参照
[論文] DNAをガイドとする高度好塩菌由来アルゴノートによるゲノム編集:Chunyu Han (河北科技大学)
- Cas9はsgRNAにガイドされて標的のDNAを切断する。アルゴノート(Argonaute, Ago)は5’末端がリン酸化された一本鎖のRNAまたはDNA(ssDNA)にガイドされてRNAまたはDNAを切断する(挿入図1右のRNAi経路参照)。Hanらは今回、高度好塩菌Natronobacterium gregoryi 由来のAgoが、CRISPR/Cas9システムよりも優れたゲノム編集ツールになり得ることを示した。
- Thermus thermophilus とPyrococcus furiosus のAgoは5’末端がリン酸化されたssDNAをガイドとしてその相補的なDNAを65°Cを超える高温で切断することが知られていた。今回、高度好塩菌のNatronobacterium gregoryi SP2のAgo(NgAgo)が37°CでヒトのdsDNAを切断することを見出した。
[情報拠点注] 挿入図2はT. thermophilus と5’末端リン酸化ssDNAとの複合体構造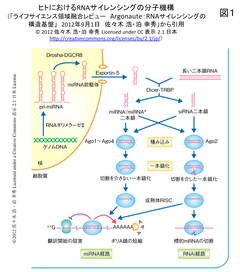

- ヒト293T細胞において、NgAgoは5’末端をリン酸化したssDNAにのみ結合し、それをガイドDNA(gDNA)として, gDNAと相補的な標的dsDNAを切断した(すなわち、ssRNAあるいは5’末端がリン酸化されていないssDNAとは結合しなかった)。
- NgAgoは内在核酸と結合せず内在核酸によるオフターゲット・サイトへの誘導が生じなかった(5’末端がリン酸化されている内在性ssDNAは極めて少ないことを示唆してもいる)。
- NgAgoをコードするプラスミドとgDNAを同時に細胞に導入した場合に限り37°CでDNA切断活性を示した。すなわち、NgAgoは発現している間にgDNAに結合する。また、いったん結合したgDNAを他の5’末端リン酸化ssDNAと置換することがなかった(‘one-guide faithful’)。
- NgAgoはdsDNAを単に切断するだけでなく数塩基を削除した。
- HeLa細胞のCMVプロモーターを標的とする比較実験で、NgAgo-gDNAは、Cas9-sgRNAと同等のゲノム編集効率を示した。この際にgDNAの最適長が24ntであることも明らかになった。NgAgo-gDNAのdsDNA切断活性は、HeLa細胞や乳がん細胞株MCF-7と白血病細胞株K562においても確認。
- DYRK1A 遺伝子のエクソン11を標的とする比較実験でもNgAgo-gDNAとCas9-sgRNAの切断効率は同等であったが、GCリッチなHBA2 とGATA4 の場合は、NgAgo-gDNAの切断効率が上回った。
- gDNAと標的のミスマッチに対する許容度については、連続した3塩基のミスマッチの場合は0%であったが、1塩基のミスマッチの場合は、20%前後であった(CRISPR/Cas9の場合は5塩基のミスマッチまで許容するとする報告がなされている)。
- 293T細胞のDYRK1A 遺伝子を標的とするDNA断片導入を11.7%の効率で実現、オフターゲットの挿入は非検出。
- NgAgoのサイズは887アミノ酸とCas9の1,368アミノ酸の3分2程度であり、標的サイトの認識にPAMを必要としない。また、gRNA(ssDNA)は、設計、合成、ならびに用量の調節が容易
コメント