[出典] "Multiplexed detection of proteins, transcriptomes, clonotypes and CRISPR perturbations in single cells" Mimitou EP [..] Smibert P. Nat Methods. 2019-04-22.
概要
[注] bioRxiv投稿版に拠るCRISPRメモ_2018/11/11第2項から引用:
- 先行研究で、Cellular Indexing of Transcriptomes and Epitopes by sequencing (CITE-seq) を開発したNew York Genome Centerなどとジャクソン研究所の共同研究チームは今回、CITE-seqにCRISPRスクリーンとCell "hashing"技術を組み合わせて、掲題にある一細胞5種類の同時測定を可能とするECCITE-seq (Expanded CRISPR-compatible CITE)へと拡張し、不均一な細胞集団からの悪性リンパ腫細胞の選別と分子マーカ同定を実現した。
背景
- この20年あまりRNAシーケンシングによる単一細胞 (scRNA-seq)のプロファイリング技術が長足の進歩を遂げてきた (Front Cell Dev Biol 2019 Figure 1引用下図左参照)。
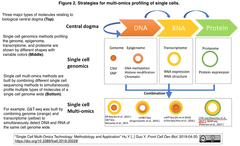
- 近年になって、scRNA-seqに、ゲノムシーンシング、クロマチン・アクセシビリティー、メチル化、免疫フェノタイピングといったハイスループット・アッセイを組み合わせた単一細胞のマルチオミックス・プロファイリングが実現された (Front Cell Dev Biol 2019 Figure 2引用上図右参照)。
- さらに、合成マーカに基づく細胞系譜解析技術との融合や、プール型CRISPRスクリーニングとscRNA-seqの融合 (CRISPRメモ_2018/02/05 1. CRISPR/Cas9遺伝子編集結果を単一細胞RNA-seq(scRNA-seq)で読み出す手法一覧 )も進んだ。
CITE-seqとCell Hashing (CITE-seqのWebサイト参照)
- New York Genome Center (NGC)とNew York University School of Medicine (NYU-M)の研究グループは2017年に、同一の単一細胞のトランスクリプトームと細胞表面タンパク質を解析可能とするCITE-seq (Cellular Indexing of Transcriptomes and Epitopes by sequencing, Nat Methods 2017)を報告した。続いて2018年に、BioLegend Incと共に、異なる細胞集団を混合して1回のCITE-seqの結果を元の細胞集団に戻して (demultiplexing)解析可能とするCell Hashing法を報告した(Genome Biol 2018 Fig.1-A引用下図参照)。

- CITE-seqもCell Hashingも抗体とDNAオリゴヌクレオチドの複合体を使用する。CITE-seqでは、抗体は単一細胞の表面タンパク質に特異的に結合する役割を担い、DNAオリゴヌクレオチドは、各抗体(ひいてはタンパク質)に特異的な分子バーコードを帯び、Drop-seqや10xGenomicsのChromium system Single Cell 3´ Solutionなどのpoly-dTプライマーを利用するscRNA-seqに読み取られる"Antibody-Derived Tags (ADT)"の役割を担っている。
- こうして、同一の単一細胞について、細胞内在mRNAから逆転写したcDNAと共に、抗体特異的DNAオリゴヌクレオチドから逆転写したADTのシーケンシングが可能になる。Cell Hashingでは、サンプル集団に特異的に遍在する表面タンパク質に対する抗体とDNAオリゴヌクレオチド (hashtag-oligo, HTO)の複合体を使用する。
ECCITE-seq
- NGC, NYU-MにJackson Laboratory for Genomic Medicineが加わった研究グループは今回、CITE-seqとCell Hashingを、10x Genomicsの5P / V(D)J single cell kit に適合させて単一細胞の免疫クロノタイプ同定を実現し、さらに、sgRNAスキャフォールドにアニールする逆転写プライマーを組み込むなどによりsgRNA配列の直接検出も実現して、CRISPRスクリーンに多様なリードアウトを柔軟に組み合わせることを可能とする ECCITE-seqに到達した。
ECCITE-seqの実証実験
- ヒト末梢血単核細胞(PBMC)集団 、2種類のT細胞リンパ腫細胞株 (MyLaとSez4)集団、マウスNIH-3T3細胞集団の混合プール (総細胞数 5,935)を対象とする1回のECCITE-seqにより、トランスクリプトーム、プロテオーム、TCRα/β、TCRγ/δ、non-targeting sgRNAsを、それぞれの細胞集団ごとに同定可能なことを示した。
- K562細胞を対象として、細胞表面マーカ (CD29とCD46)と細胞内シグナル伝達分子(JAK1とp53)それぞれを標的とするsgRNAsおよびコントロール用の標的無しsgRNAs2種類を送達し、ECCITE-seqにより同一細胞でのmRNAのレベルとタンパク質のレベルを比較した結果、sgRNAによる遺伝子変異の影響を見るにmRNAレベルよりもタンパク質レベルが高感度であるとした。
- マーカ49種類の抗体パネルを利用したECCITE-seqにより、健常者由来のと皮膚T細胞リンパ腫 (cutaneous T-cell lymphoma, CTCL)患者由来のPBMC (細胞数それぞれ3,733と3,866)について、1回のECCITE-seqにより、トランスクリプトーム、マーカタンパク質ならびにクロノタイプの比較も行なった。

コメント