[出典] Stacia K. Wyman, Aram Avila-Herrera, Stephen Nayfach, Katherine S. Pollard. “A most wanted list of conserved protein families with no known domains” bioRxiv. Posted October 23, 2017.
- ゲノムシーケンシングとメタゲノミクス由来配列データ拡大のペースに、遺伝子機能解析のペースは大きく遅れを取っている。配列相同性に基づいたアノテーションから、配列データ由来のタンパク質コーディング遺伝子とRNA遺伝子のドメインと機能を推定可能であるが、しばしば、実験的裏付けのあるドメインまたは遺伝子ファミリーへの相同性が低い故に機能を推定できないままになる推定遺伝子が現れる。
- Katherine S. PollardらGladstone Instituteの研究チームは、この現象を定量化し、機能解析の優先順位をつけるために、バイオインフォマティクス・パイプラインを開発し、配列データベースに内在する新奇機能遺伝子の広がりと、優先的に機能解析すべき遺伝子のリスト”most wanted list of genes to characterize”の作成法を提示した。
SFamsからの出発
- SFamsは、2,928ゲノム由来の~1,050万件のタンパク質配列から、ペアワイズBLASTPからの配列一致度をもとにしたマルコフクラスタリングを核とするワークフロー(下図参照)により全自動で構築されたタンパク質ファミリーデータベースである。今回、SFamsに登録されていた345,641タンパク質ファミリーから、ファミリー内のユニークなタンパク質配列が2種類以下のファミリーと、50%を超えるタンパク質配列のスタートコドンまたはストップコドンを欠いているファミリーを捨て、残った224,409全長タンパク質ファミリーを対象として解析を始めた。
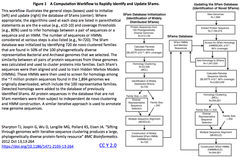
PFamとNCBI Conserved Domain Database(CDD)による新奇ドメイン判定
- SFams配列全長にわたり完全一致する配列を検索し、PFamまたはCDDにおいてアノテーションが付されているドメインを含まないファミリー118,607種類を見出した。参照にあたり相同性検索を使用しなかったのは、SFamsの元データであったゲノム配列由来のタンパク質配列は、相同性を介したアノテーションが、PFamとCDDには取り込み済みと判断したからである。
NCBI Taxonomyに拠る生物種間分布の判定とUniProt参照
- 絞り込んだタンパク質ファミリーの各配列の由来生物種をNCBI Taxonomyで特定し、種、属、科、目、綱(class)、目、界およびドメイン間のファミリーの分布を分析した(下図参照)。その結果から、少なくとも2種類以上の綱からの配列を含むタンパク質ファミリー6,668種類を見出し、FUnkFams(Function Unknown Families)と命名した。FUnkFamsの84.3%は、UniProtに含まれず、UniProtに含まれていた場合もそのアノテーションの多くが”hypothetical”または”uncharacterized”であった。

- Tara Oceans Expedition(TO: 20箇所210種類の生態系由来243サンプルのデータ;深度依存と年変動)とHuman Microbiome Project(HMP: 健常者300人の口腔、気道、皮膚、腸、膣由来の699サンプルのデータ)における分布を分析した(下図参照)。FUnkFamsの56.5%が942メタゲノムのいずれかに見出され、32.5%が複数のメタゲノムで見出されたが、TOとHMPの双方に見出されたFUnkFamsは比較的少数(13.3%)であった。
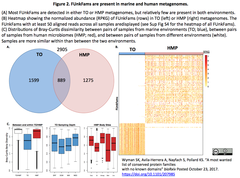
FUnkFamsの分布とサンプルの特性との相関
- TOにおいて硝酸塩のレベルと強く相関するFUnkFamsが3種類存在した。そのうち1種類は塩分濃度と経度とも強く相関し、その他は経度、緯度、温度および深度と強く相関した。HMPにおいては、FUnkFamsは由来部位と相関し、BMIや喫煙あるいは食事との相関は弱かった。こうした分析から、FUnkFamsの機能を推定していくことが可能である。
コメント