1.CADS:CRISPR/Cas12aを利用して、DNAにコードされている情報を安全に保持し伝達するDNAステガノグラフィー(データ隠蔽技術)
- "CADS: CRISPR/Cas12a-Assisted DNA Steganography for Securing the Storage and Transfer of DNA-encoded Information". Li S, Liu JK, Zhao G, Wang J. ACS Synth Biol. 2018 Mar 29.
- 古典的DNAステガノグラフィーは、"秘密なメッセージ"を膨大なジャンクDNAに埋め込み、特定のプライマーを鍵としてPCRで読み出し、DNAシーケンシングを経て解読する手法であり、プライマーの漏洩が弱点になっていた。上海の研究チームは今回その弱点を克服するために、鍵となるプライマー(トルー・キー)と非特異的プライマー(フェイク・キー)と混合するか、または、鍵となるプライマーの3’末端に冗長な配列を融合することで、"pre-key"を開発した。CRISPR/Cas12aのssDNAトランス切断活性を利用して、フェイク・キーからトルー・キーを生成し、解読へ進む。
2.[特許]ゼノ核酸を利用したsgRNAによるゲノム編集の効率向上
- “SPECIFIC SYNTHETIC CHIMERIC XENONUCLEIC ACID GUIDE RNA; s(XNA-gRNA) FOR ENHANCING CRISPR MEDIATED GENOME EDITING EFFICIENCY” US 201/0066258 A1. PubDate 03/08/2018. Invent/Assignee Powell, Michael J.
3.In vivoで精密な遺伝子ノックインを可能とするHMEJ-based CRISPR/Cas9システム(ビデオジャーナル)
- ”CRISPR/Cas9-mediated Targeted Integration In Vivo Using a Homology-mediated End Joining-based Strategy” Yao X, Wang X, Liu J, Shi L, Huang P, Yang H. J Vis Exp. 2018 Mar 12;(133).
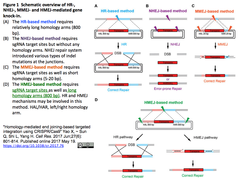
- CRISPR/Cas9は遺伝子導入の強力なツールであるが、HRの効率が低い非分裂細胞への適用は課題である。HMEJ-based CRISPR/Cas9は、挿入配列と一対の長い相同アーム(800 bp)に加えて、ゲノムの挿入標的サイトを特定するsgRNAで切断される配列を帯びたベクターを利用する (下図 Figure 1-D参照(*))。
- Yang Hらはこの手法をCell Research (2017)(*)に発表し、マウスとサルの胚、肝細胞、神経細胞におけるノックインを、HR-、NHEJ-およびMMEJ-よりも高効率で実現
- (*)"Homology-mediated end joining-based targeted integration using CRISPR/Cas9" Yao X, ~ Sun Q, Shi L, Yang H. Cell Res. 2017 Jun;27(6):801-814. Published online 2017 May 19.
4.CRISPcut:ヒト細胞におけるCRISPR/Cas9実験に向けた最適なsgRNAsを設計するツール
- "CRISPcut: A novel tool for designing optimal sgRNAs for CRISPR/Cas9 based experiments in human cells" Dhanjal JK, Radhakrishnan N, Sundar D. Genomics. Available online 29 March 2018.
- オフターゲットに関するこれまでの知見を生かした上で、細胞型ごとに異なるクロマチンアクセシビリティを組み込んでオープンなクロマチン領域を標的とするsgRNAsを選択するCRISPcutを開発
- Webサイト:http://web.iitd.ac.in/crispcut/
5.精神疾患に相関する脆弱X症候群関連タンパク質Fxr1とその負の調節因子グリコーゲン合成酵素キナーゼ3β (Gsk3β)は、不安とグルタミン酸作動性神経伝達の調節因子である
- "Mental illnesses associated Fxr1 and its negative regulator Gsk3β are modulators of anxiety and glutamatergic neurotransmission" Khlghatyan J, ~ Beaulieu M. Front Mol Neurosci. Accepted: 28 Mar 2018.
- Fxr1とGsk3βは、数種類の向精神薬共通の標的であり、GSK3bの多型とGsk3βの機能的関連も示唆されている。カナダの研究チームは今回、成体マウス脳において、CRISPR/Cas9によるノックアウトと過剰発現から、両者の相互作用と分子機構を明らかにした。
6.[レビュー]HIV持続感染に取り組む:"shock and kill"療法には薬理かCRISPR技術か
- "Tackling HIV Persistence: Pharmacological versus CRISPR-Based Shock Strategies" Viruses. 2018, 10(4), 157Darcis G, Das AT, Berkhout B. .
- HIVが潜伏している細胞(リザーバー細胞)がHIVの完全寛解を阻んでいるが、リバーサー細胞中のHIVを活性化した上でHIV産生細胞を破壊する療法が模索されている。HIVが宿主細胞に潜在する分子機構、"shock"を与える薬剤およびCRISPR技術による"shock"戦略をレビュー (下図参照)。
7.[展望]CRISPRにおける腎臓の遺伝子編集
- "CRISPR Gene Editing in the Kidney" Cruz NM, Freedman BS. Am J Kidney Dis. Available online 30 March 2018.
- CRISPR技術の腎疾患の研究と治療への応用:多能性細胞由来の腎オルガノイドの腎疾患モデルから多発性嚢胞腎疾患とネフローゼ症候群の細胞メカニズムについて新たな知見;腎癌と免疫疾患のCRISPRによる細胞療法治験開始;CRISPRでウイルスを除去したブタからの腎移植研究;課題(腎臓へのCRISPRシステムの送達;ゲノムにおける遺伝子編集の調節)
8.[OPINION]CRISPR in vivoスクリーンによる癌研究と癌療法の現状と展望
- "Cancer CRISPR Screens In Vivo" Chow RD, ChenS. Trends Cancer. Available online 30 March 2018.
- これまでCRISPRスクリーンはほとんどin vitroで行われてきたが、最近、ヒト癌モデル動物におけるin vivoスクリーンが行われ腫瘍微小環境におけるドライバー遺伝子が同定された。また、完全な免疫応答性を備えた実験動物にCRISPR技術で多重変異を導入することで、個別化医療に有用な患者特異的モデルが作出された。
9.[FORUM]植物RNA編集ツールとしてのCRISPR/Cas13
- "CRISPR/Cas13 as a Tool for RNA Interference" Ali Z, Mahas A, Mahfouz M. Trends Plant Sci. 2018 Mar 28.
- 植物におけるCRISPR/Cas13の機能解析の必要性や期待できる応用について考察
10.高効率なCas9遺伝子編集技術による大麦におけるMORC1の機能解析
- "Further analysis of barley MORC1 using a highly efficient RNA-guided Cas9 gene editing system" Kumar N, Galli M, Ordon J, Stuttmann J, Kogel KH, Imani J. Plant Biotechnol J. 2018 Mar 25.
- MORC遺伝子の穀物における機能の解析は、ノックアウト (KO)系統が存在せず、また、MORCが遺伝子サイレンシングに関与するためRNAiの適用にも問題があり、困難であった。
- 大麦MORC遺伝子ファミリーの一種であるHvMORC1を、大麦の新奇RNA Pol Ⅲ依存性U3 snRNAプロモーターでsgRNAを発現させることで、SpCas9による高効率編集(T0世代で77%; T1世代で70-100%)を実現し、シロイヌナズナのMORCと対照的に、大麦のノックアウト系統では病原真菌に対する耐性が向上することを見出した。
11.野生イチゴFragaria vescaの効率的ゲノム編集
- "Efficient genome-editing of wild strawberry genes, vector development, and validation" Zhou J, Wang G, Liu Z. Plant Biotechnol J. 2018 Mar 25.
- これまで事例が少なかった園芸植物の高効率なゲノム編集を、シロイヌナズナのU6プロモーターAtU6-26または野生イチゴU6プロモーターFveU6-2によるsgRNA発現で、実現。
12.CRISPR-Cas9によるClostridium difficileのゲノム編集
- "Genome engineering of Clostridium difficile using the CRISPR-Cas9 system" Wang S, ~ Wang Y. Clin Microbiol Infect. 2018 Mar 28.
- C. difficileのspo0Aの削除を効率100%で、嫌気性GFP遺伝子のノックインを効率80%で実現
13.CRISPR/Cas9による極限環境微生物Halomonas sppのゲノム編集
- "CRISPR/Cas9 Editing Genome of Extremophile Halomonas spp" Qin Q, ~ Guo Y, Chen GQ. Metab Eng. Available online 30 March 2018.
- 次世代の工業微生物として期待されているHalomonasのゲノム編集はこれまで、極めて労力と時間を要する自殺遺伝子発現プラスミドを利用した2段階の相同組換えによって行われてきた。清華大学の研究チームは今回、CRISPR/Cas9によって、4.5-kbまでのDNAの削除とゲノムへの挿入を3週間以内に実現。また、8種類の変異体にもとづいて4種類の遺伝子がグルコース異化作用に与える影響を解析
14.[レビュー]CRISPR-Cas技術による遺伝子操作が困難であった微生物のゲノム編集の進展
- "CRISPR-based genomic tools for the manipulation of genetically intractable microorganisms" Shapiro RS, Chavez A, Collins JJ. Nat Rev Microbiol. 2018 Mar 29.
- 相同組換え (HR)の頻度が低くまた効率的な組換えに長い相同シーケンスを必要とすることから、マイクバクテリウム、菌類および病原性微生物のHRをベースとした遺伝子操作ひいては逆遺伝学の展開が遅れていたが、CRISPR技術によって古典的なHRの手法を回避することが可能になった:ゲノムワイドな機能喪失スクリーン;遺伝子発現抑制;遺伝子削除;遺伝子ノックイン;遺伝子多重編集から遺伝子間相互作用解析へ;産業微生物のゲノム工学;展望

コメント