[出典] "Pooled CRISPR screens with imaging on microraft arrays reveals stress granule-regulatory factors" Wheeler EC, Vu AQ [..] Yeo GW. Nat Methods 2020-05-11.
[マイクロラフト参考論文]
UCSDとU North Carolina at Chapel Hillの研究グループが、プール型sgRNAsライブラリに基づくCRISPR-Cas9スクリーンに、マイクロラフト (microraft)技術と、深層学習モデルに基づく自動化ハイコンテント・イメージング技術を組合せることで、RNA結合タンパク質 (RBP) の機能同定を実現し、この新手法をCRaft-ID (CRISPR-based microRaft followed by guide RNA IDentification)として報告した。
- 20セットのマイクロラフト上で、RBSs (> 1,000)とアノテーションされている遺伝子群を標的とするsgRNAs (>12,000)ライブラリーに基づくプール型CRISPR-Cas9スクリーンにて、クローン細胞 (~ 5-20個)のコロニーを分離培養し、119,050種類のクローンに、ヒ酸ナトリウムでストレスを加え、共焦点顕微鏡に基づくハイコンテント・イメージングを介して、CRISPRノックアウトによってストレス顆粒レベルの低下をもたらしたsgRNA (ひいてはRBP)を同定した [原論文 Fig. 1: Microraft arrays enable the culture and stress granule quantification of thousands of clonal cells 参照]。
- イメージ判定の自動化は、畳み込みニューラルネットワーク (CNN)モデルを構築することで実現した。
- CRaft-IDによって、既知のストレス顆粒調節因子6種類を同定し、新たに、その欠損によってストレス顆粒レベルが低下する下RBSsを17種類同定した。
[マイクロラフト参考論文]
- Micromolded Arrays for Separation of Adherent Cells. Wang Y [..] Allbritton B. Lab Chip 2020-11-07 (PMC ID)
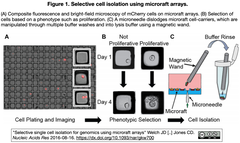
- "Selective single cell isolation for genomics using microraft arrays" Welch JD [..] Jones CD. Nucleic Acids Res 2016-08-16. [Figure 1引用下図参照]
コメント