[出典] "CRISPR-CBEI: a Designing and Analyzing Tool Kit for Cytosine Base Editor-Mediated Gene Inactivation" Yu H, Wu Z, Chen X, Ji Q, Tao S. mSystems 2020-09-22.
[注] CBEI (cytosine base editor-mediated gene inactivation)
西北農林科技大学と上海科技大学の研究チームは、BE3 [APOBEC1-nCas9-UGI]をはじめとする各種CBEを利用して、CAA, CAG, CGA, あるいはTGGをストップコドンへ変換することで、標的遺伝子の不活性化を実現するにあたり、CRISPR-Cas9によるノックアウト用のsgRNAよりも複雑な条件を勘案する必要があるCBE用のsgRNAs設計を支援するWebツール [*1]とPythonによるコマンドラインのツール"autocbei" [*2]を開発・公開した。
[*2] Github (https://github.com/atlasbioinfo/CRISPR-CBEI/blob/master/autocbei/README.md); PyPI (https://pypi.org/project/autocbei/); Anaconda ()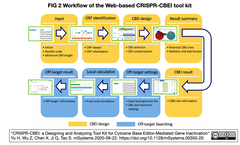

Webツール上では、FIG. 2から引用した右図にあるように、配列入力から始まり, ORFs同定、CBEの選択[*3]とカスタム化, CEBI候補サイトとその分布などの解析結果, およびCBEIの予測結果、続いて、ローカルファイル上の配列を対象とした高速な[*4]オフターゲット解析とその結果の出力に至る。sgRNAs候補探索にあたっては、スペーサの長さ、PAMの配列と位置、CBEのウインドウ幅、および、探索範囲を指定可能である。
 [*3] 評価したCBEs: BE-PLUS, BE, Cas12a-BE, EE-BE3, Sa(KKH)-BE3, SaBE, Target-AID-NG, Target-AID, VQR-BE3, VRER-BE3, xBE3, YE1-BE3,およびYEE-BE3 [右図に、大腸菌からヒト細胞株まで12種類のCBEに基づくCBEIの解析結果をFIG4から引用]
[*3] 評価したCBEs: BE-PLUS, BE, Cas12a-BE, EE-BE3, Sa(KKH)-BE3, SaBE, Target-AID-NG, Target-AID, VQR-BE3, VRER-BE3, xBE3, YE1-BE3,およびYEE-BE3 [右図に、大腸菌からヒト細胞株まで12種類のCBEに基づくCBEIの解析結果をFIG4から引用][*4] i5-3470/3.20GHzのCPUのコンピュータでヒトゲノム全長 (~3 Gb)を対象とするオフターゲット予測に要した時間は~5分
コメント