[注] ハイブリダイゼーション-近接依存性標識 (Hybridization-proximity labeling, HyPro標識法)
[出典] "Hybridization-proximity labeling reveals spatially ordered interactions of nuclear RNA compartments" Yap K, Chung TH, Makeyev EV. Mol Cell. 2021-11-05. https://doi.org/10.1016/j.molcel.2021.10.009
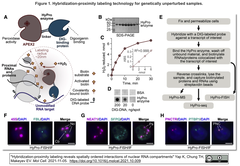 King’s College Londonの研究チームは,APEX2とジゴキシゲニン (DIG)結合ドメインを柔軟なリンカーで連結したHyPro組み換え酵素 [挿入図 Aの上段を参照]を作出・利用することで,以下のHy-Proのワークフローを実現した [挿入図Aの下段と挿入図E参照]:
King’s College Londonの研究チームは,APEX2とジゴキシゲニン (DIG)結合ドメインを柔軟なリンカーで連結したHyPro組み換え酵素 [挿入図 Aの上段を参照]を作出・利用することで,以下のHy-Proのワークフローを実現した [挿入図Aの下段と挿入図E参照]:- 細胞の固定化と透過処理
- DIG標識したDNAプローブと関心のある転写物(以下,標的RNA)のハイブリダイゼーション
- HyPro酵素のDIG結合ドメインが,DIG標識DNAプローブを介して標的RNAに係留され,HyPro酵素のAPEXが過酸化水素 (H2O2)存在下でその近傍の一連のRNAとタンパク質をビオチン化することで,
- リバースクロスリンクと細胞溶解を経て,ビオチン化されたタンパク質とRNAをストレプトアビジンビーズで捕捉する.
- 共局在していたタンパク質とRNAsをそれぞれ質量分析 (HyPro-MS)とディープシーケンシング (Hydro-seq)によって同定する.また,FISH (Hydro-FISH)によって,DIG標識プローブの特異性を評価する.
研究チームは,HyProを利用して,発現レベルが著しく異なるノンコーディングRNAである45S,NEAT1,およびPNCTRについて,既知および未知の空間的近接関係を明らかにした.注目すべきは,アデノシンからイノシンに編集された不完全に処理された転写産物の多くが,それらがコードされている染色体領域と,NEAT1を含むパラスペックル・コンパートメントとの間のインターフェースに蓄積されていたことである.これらのターゲットのうち少なくともいくつかは,その最適な発現のためにNEAT1を必要としている.
本研究は,多様な生物医学的コンテクストにおけるRNAインタラクトームを解析するための汎用的なツールキットを提供するとともに,哺乳類細胞における核の機能的構造についての理解を深めた.
コメント