[注] peCHYRON: prime editing Cell HistorY Recording by Ordered iNsertion
[出典] "Molecular recording of sequential cellular events into DNA" Loveless TB, Carlson CK, Hu VJ, Dentzel Helmy CA, Liang G [..] Liu CC. bioRxiv. 2021-11-07 [プレプリント]. https://doi.org/10.1101/2021.11.05.467507
UC Irvineの研究グループは先行研究で,Cas9ヌクレアーゼとhgRNA (homing guide RNA, Nat Methods 2017),およびDNA書き込み因子を利用して,単一の遺伝子座 (CHYRON遺伝子座)に時系列に応じて挿入変異を蓄積し、それを次世代シーケンシングで読み出し、解析することで、細胞系譜の追跡 (lineage tracer)と細胞ストレスの遡及(stimulus recorder)を実現するCHYRON (Cell HistorY Recording by Ordered iNsertion)法を開発 [*]していたが,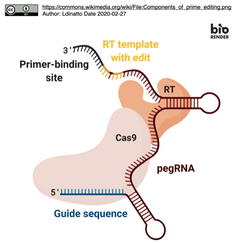 今回,プライムエディティング [右図参照]のPE2をベースとするCHYRONを,peCHYRONとしてbioRxiv に投稿した.
今回,プライムエディティング [右図参照]のPE2をベースとするCHYRONを,peCHYRONとしてbioRxiv に投稿した.
 今回,プライムエディティング [右図参照]のPE2をベースとするCHYRONを,peCHYRONとしてbioRxiv に投稿した.
今回,プライムエディティング [右図参照]のPE2をベースとするCHYRONを,peCHYRONとしてbioRxiv に投稿した.- DNAレコーダーは,一過性の生物学的事象を非侵襲的に細胞内のゲノム上に保存されていく変異へと変換することで,後に,ハイスループットDNAシーケンシングを用いて,事象を時系列に応じて再構築可能とするシステムである.
- これまでに開発された種々のDNAレコーダーが,多量の情報の記録,耐久性のある記録,複数のタイムスケールでの長時間記録,利用者が選択した複数の信号の多重記録,時間分解された信号の記録を実現してきたが,すべてを同時に実現した例はなかったところ,peCHYRONはそれを実現した.
- peCHYRONは,その逆転写酵素(RT)の鋳型配列が,最大6ビットの情報をコードする可変3ntの配列 (以下,シグネチャー変異)と,次の挿入ステップのターゲット配列となる17ntの長さの一定の伝播配列 (constant propagation sequence)配列を挿入するようにプログラムされている.こうすることで,挿入の1サイクル後,直前のプロパゲータ配列はPAM配列から遠位になりしたがって不活性となり,新しい20bpのターゲットサイトを形成する新しいプロパゲータ配列がPAMに隣接することで活性となる.次の編集ステップで,次のプロパゲータ配列とともに,新たな可変3-nt配列が挿入されることを無限に繰り返す.このようにして,細胞事象が順次シグネチャー変異として記録されていく.
- トリプレットDNA (3nt)のシグネチャー変異は,理論的には64種類のイベントを記録可能であり,複雑な細胞事象を記録可能であり,17ntの伝播配列が,一連のシグネチャー変異の区切りとして機能する.なお,この戦略の実装にあたっては,2種類の伝搬配列を交互に利用する必要があった [Figure 1参照 ].
- peCHYRONでは,欠失の危険性のあるDNA二重鎖切断 (DSB)を回避するPE2を使用していることから,蓄積された挿入は編集継続中も破壊されることがない.
- peCHRONの概念実証実験では,少なくとも41種類のpegRNAを利用した細胞系譜再構成が可能であり,また,異なるpegRNAを誘導性プロモーターを介してパルス状に発現させることにより,パルス・シーケンスを再構築し,これを生体内外からの刺激と結合させることにより,多重事象を時間分解可能な形で記録する可能性を示した.
[*]
- [20210323更新] 細胞事象DNAレコーダに、挿入変異を累積する新手法CHYRON 加わる.https://crisp-bio.blog.jp/archives/17700071.html; "Lineage tracing and analog recording in mammalian cells by single-site DNA writing" Loveless TB [..] Liu CC. Nat Chem Biol. 2021-03-22. https://doi.org/10.1038/s41589-021-00769-8
コメント